AI for Science新突破:2024年4月12日,复旦大学计算机科学技术学院颜波教授团队在Nature Methods杂志上在线发表了题为“Pre-training a Foundation Model for Generalizable Fluorescence Microscopy-Based Image Restoration”的研究论文。

背景介绍
荧光显微镜是研究生命科学中细胞和组织发育时空动态的不可或缺工具,能够以高分辨率对细胞内的生物过程进行体成像。然而,其成像质量受显微镜光学和生物样本光敏感性的限制。荧光显微镜图像重构可以突破成像速度、空间分辨率、曝光量和成像深度之间的物理约束,从而更好地揭示细胞内部和生物分子的复杂交互作用和动态变化规律,扩展生物现象的观察和研究范围,有力推动基础科学的发展,也为疾病诊断和新药开发等应用性研究提供重要工具。
在本文中,研究者构建了首个统一的荧光显微镜图像增强基础模型(unified foundation model for fluorescence microscopy-based image restoration, UniFMIR),该模型可以解决不同类型的荧光显微镜图像增强问题,包括图像超分辨率、各向同性重构、3D去噪、图像投影和过程重建,在跨模态数据集和跨任务实验上表现出优异的泛化性和通用性,显著提升图像增强精度,多维度突破荧光显微成像极限,揭示清晰的活样本细胞结构。论文将基础模型应用于荧光显微镜图像增强领域,可为研究人员进一步开发荧光显微镜图像智能增强方法提供有益启发。
UniFMIR框架概述
UniFMIR是一个解决不同类型的荧光显微镜图像增强问题的预训练模型,其主要网络架构和工作流程如图1所示。针对荧光成像模式多样、降质类型复杂和增强过程迥异等研究难点,研究者统一了不同成像模式、降质类型、增强任务,并为不同任务设计不同结构的特征提取和图像重构网络。UniFMIR采用了基于Swin Transformer[1]结构的特征增强模块来增强特征表示,针对不同任务的网络流程共享相同的特征增强计算。通过收集的大规模数据集对模型进行预训练,并使用不同图像增强任务的数据微调模型参数,UniFMIR展现出比专有模型更好的增强性能和泛化性。
针对Swin Transformer架构存在的模型参数量大导致部分任务推理速度慢的问题,研究了量化、剪枝等模型加速技术,提升其推理速度。针对输入图像尺度多样造成的重构结果差异问题,提出了像素矫正的数据预处理模块,保证模型重构结果对不同尺度输入的一致性。
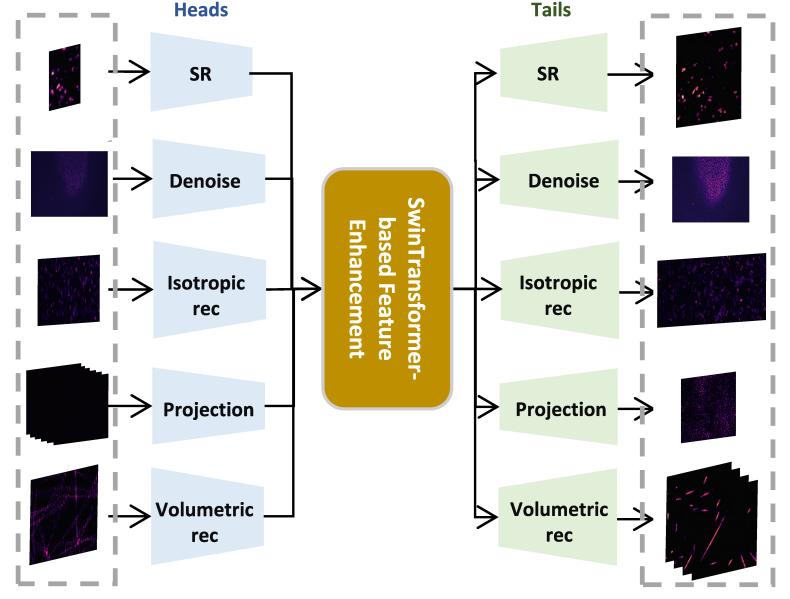
图1 荧光显微镜图像增强基础模型(UniFMIR)架构
实验结果
研究使用现有公开荧光显微镜数据来评估UniFMIR,在图像超分辨率重构、各向同性重构、3D去噪、图像投影和过程重建五个任务上的性能。
超分辨率重构:可以克服活细胞成像的理论空间分辨率限制,提升科学显微镜图像的分辨率。图2展示了在通过多模态结构照明显微镜(SIM)获得的BioSR数据集[2]上,UniFMIR对于结构复杂性不同的亚细胞结构可以高保真地恢复f-肌动蛋白(F-actin)的清晰结构细节。
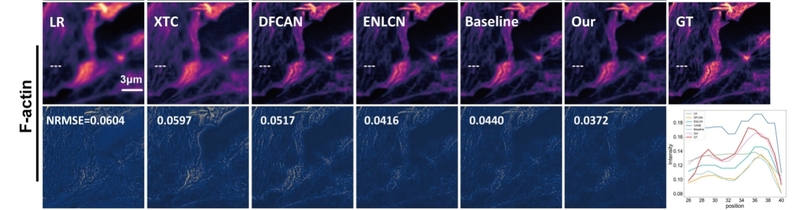
图2 UniFMIR的超分辨率重构任务性能展示
各向同性重构:可以提升荧光显微镜图像的各向同性分辨率,图3展示了UniFMIR应用于来自体积小鼠肝脏成像的各向异性数据[3](轴向分辨率比横向分辨率低10倍),可以提高轴向分辨率以提供近各向同性成像,显著提升生物样品形状的测量精度。
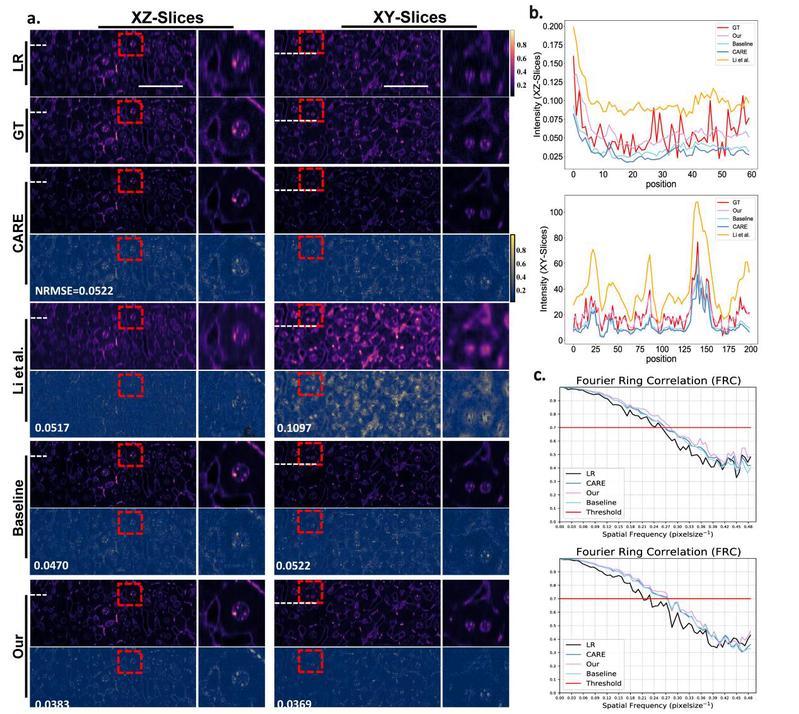
图3 UniFMIR的各向同性重构任务性能展示
3D去噪:通过计算提高荧光显微镜图像信噪比,降低高激光功率和长曝光时间对于生物样本的毒害副作用。图4展示了针对Planaria和Tribolium数据集[3]中地中海圆头涡虫和赤拟谷盗3D图像的去噪结果。

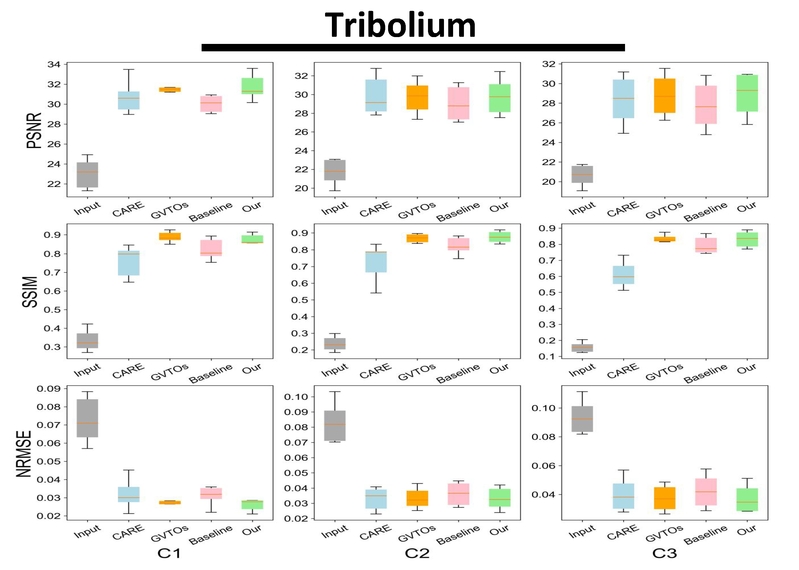
图4 UniFMIR的3D去噪任务性能展示
图像投影:可将3D立体成像投影到2D表面图像中,有助于分析和研究发育中果蝇的细胞行为。图5展示了不同方法对于C2激光功率条件Flywing数据集[3]的表面投影能力,UniFMIR在NRMSE指标上取得更高的重构精度。

图5 UniFMIR的表面投影任务性能展示
过程重建:可从2D信息构建具有均匀空间分辨率的无伪影3D图像序列,对于快速生物过程的即时成像具有重要意义。图6展示了UniFMIR可以从2D光场显微镜图像[4]中预测3D动态过程,准确重构成像对象的运动轨迹,有助于研究各种亚细胞结构和复杂的细胞动力学规律。

图6 UniFMIR的过程重建任务性能展示
结论与展望
UniFMIR为荧光显微镜图像增强提供了一个通用的解决方案,预训练的UniFMIR通过简单的参数微调便可应用于不同任务、成像模式和生物结构,展示了基础模型方法对生物成像研究的巨大推动作用。在未来工作中,可通过进一步扩展训练数据的数据量和丰富度来不断强化UniFMIR的图像重构能力。
该工作由复旦大学计算机科学技术学院数字媒体实验室完成(https://dml.fudan.edu.cn/)。实验室博士后马晨曦博士和青年研究员谭伟敏博士为该工作的共同第一作者,颜波教授为通讯作者,其他作者还包括实验室博士生何瑞安。该研究工作得到国家自然科学基金委和上海市科委项目资助。
[1] Liu, Z. et al. Swin transformer: Hierarchical vision transformer using shifted windows. In IEEE/CVF Conference on Computer Vision and Pattern Recognition (CVPR) (2021).
[2] Qiao, C. et al. Evaluation and development of deep neural networks for image superresolution in optical microscopy. Nature Methods 1–9 (2021).
[3] Weigert, S. et al. M. Content-aware image restoration: pushing the limits of fluorescence microscopy. Nature Methods 15, 1090–1097 (2018).
[4] Wang, Z. et al. Real-time volumetric reconstruction of biological dynamics with light-field microscopy and deep learning. Nature Methods 18, 551–556 (2021).
原文链接:https://www.nature.com/articles/s41592-024-02244-3
Code:https://github.com/cxm12/UNiFMIR
